Экспорт из Микроб-2 в AMRcloud
Руководство «Экспорт из Микроб-2 в AMRcloud» рассказывает о том, как имеющиеся у вас данные из программы Микроб-2 подготовить для загрузки в онлайн-платформу AMRcloud.
Создание файла
Файл экспорта представляет собой текстовый файл, использующий в качестве разделителя полей символ табуляции и имеющий кодировку WIN1251. Формирование файла выполняется на основании данных, входящих в предварительно созданный список отобранных анализов.
Для создания списка отобранных анализов вызовите команду меню Журнал > Параметры отбора:
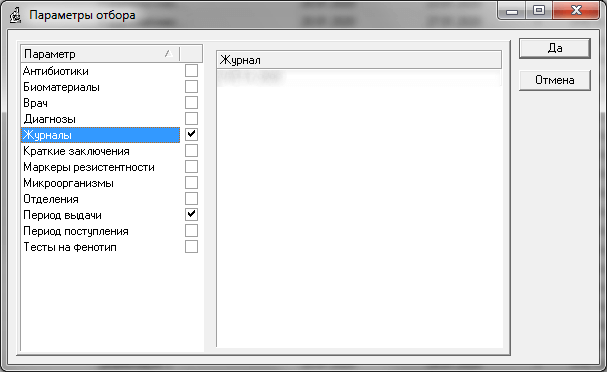
Формирование списка осуществляется путем задания следующих параметров отбора:
- Период поступления/выдачи анализов, возможные значения – весь период (от времени поступления/выдачи первого анализа до времени внесения/выдачи последнего) или определенный Пользователем;
- Отделения, возможные значения — список отделений;
- Диагнозы, возможные значения — список диагнозов, возможно указание группы диагнозов;
- Биоматериалы, возможные значения — список биоматериалов;
- Микроорганизмы, возможные значения — список микроорганизмов, возможно указание группы микроорганизмов;
- Антибиотики, возможные значения — список антибиотиков, для отбора всех указанных антибиотиков вместе - признак «только все вместе». Установка параметров отбора по антибиотикам задается отдельно по названию антибиотиков с указанием степени чувствительности.
Каждый из анализов, отобранных программой, удовлетворяет всем заданным параметрам. Как минимум, следует задать название журнала и период выдачи. Задание параметра «Дата выдачи» определяет, что в отобранный список анализов попадут только «готовые» анализы (анализы имеющие дату выдачи). Как правило, для мониторинга требуются данные именно «готовых» анализов.
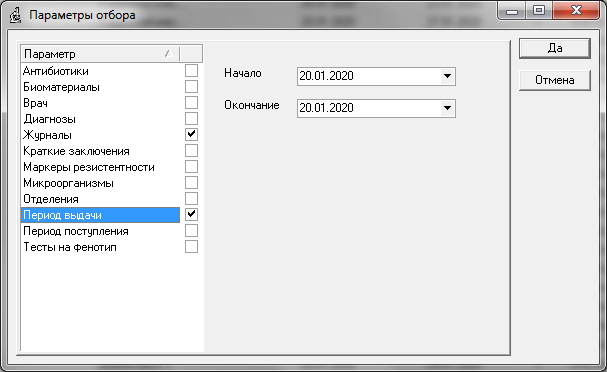
После завершения установки параметров нажмите Да для формирования Списка отобранных анализов:
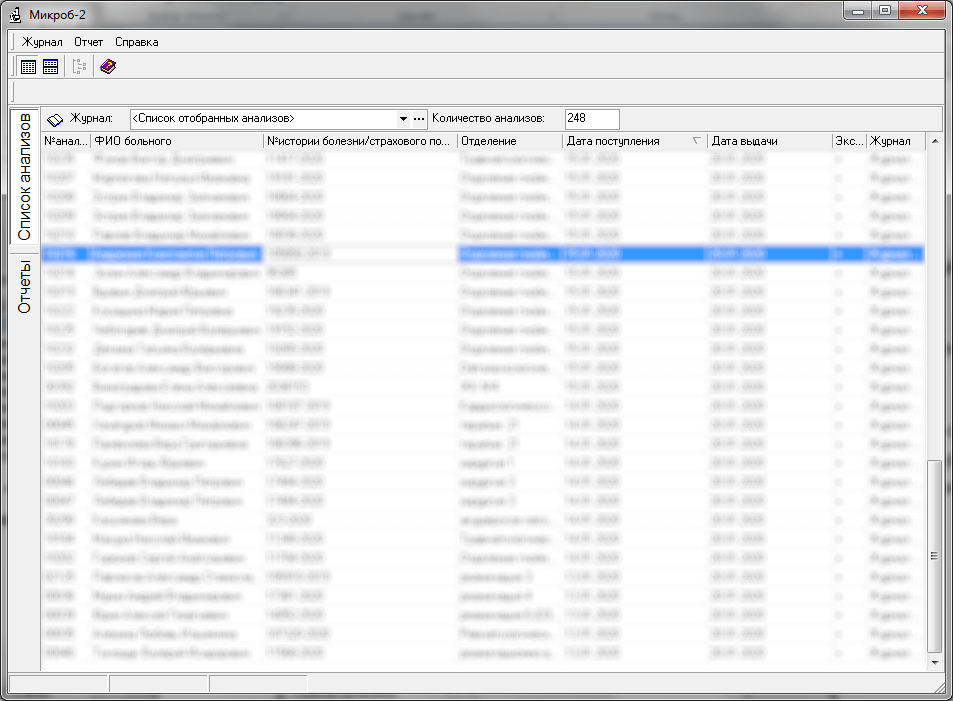
Для формирования файла вызовите контекстное меню кликнув правой клавишей мыши по списку отобранных анализов и выберите Экспорт данных для мониторинга.
Задайте имя файла и нажмите кнопку Сохранить.
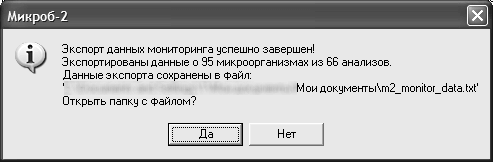
Начнется процесс экспорта данных, по окончании которого появится сообщение о его завершении. Пример такого сообщения показан на рисунке.
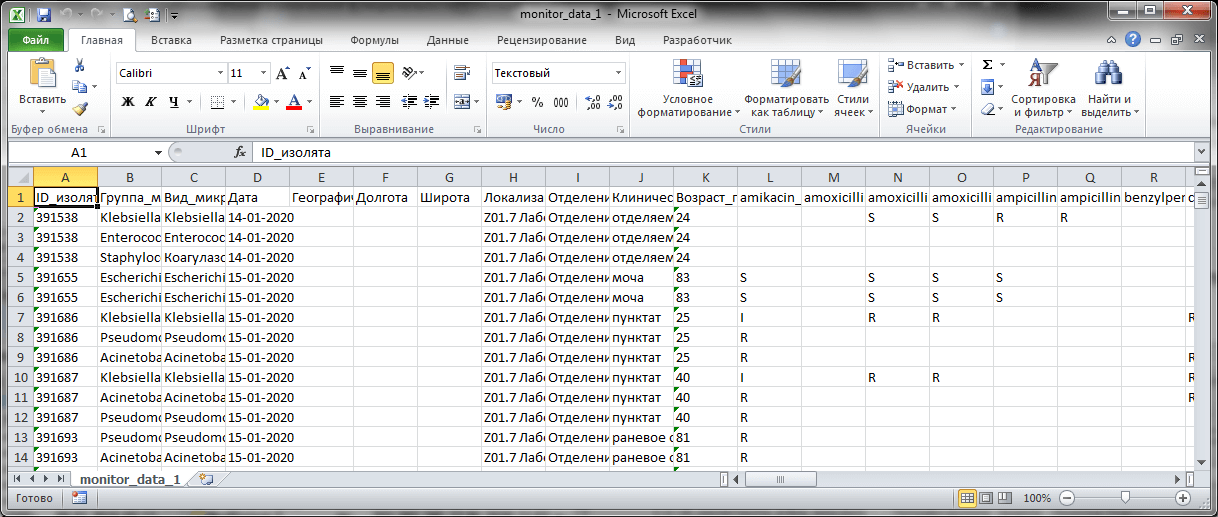
Пример созданного файла, загруженного в программу MS Excel, показан на рисунке.
Для импорта файла в MS Excel воспользуйтесь меню Данные, разделом Получение внешних данных и кнопкой Из текста. Выберите созданный файл и при импорте укажите формат файла: «1251 : Кириллица (Windows)», символ-разделитель: «Знак табуляции». Формат данных столбцов следует указывать как «текстовый», иначе некоторые значения могут быть автоматически преобразованы, например, значение МПК «4/4» может быть преобразовано в дату «04-апр».
Настройка экспорта
Обычно настройки экспорта данных мониторинга в формат AMRcloud предустановлены и не требуют дополнительной настройки, однако при наличии «пользовательских» значений Микроб-2 может потребоваться их дополнительная кодировка. Поэтому перед экспортом данных рекомендуется посмотреть и уточнить значения сопоставления в настройках программы.
Вызов настроек программы выполняется через команду основного меню Журнал > Настройки. Настройки программы индивидуальны для каждого компьютера: настройка того или иного параметра «работает» только для данного компьютера. Настройки, влияющие на создание файла для AMRcloud находятся на вкладке Отчеты и доступны через кнопку Настройки экспорта данных для мониторинга.
Общие настройки
Глобальные параметры формирования файла AMRcloud можно задать на вкладке Общие.
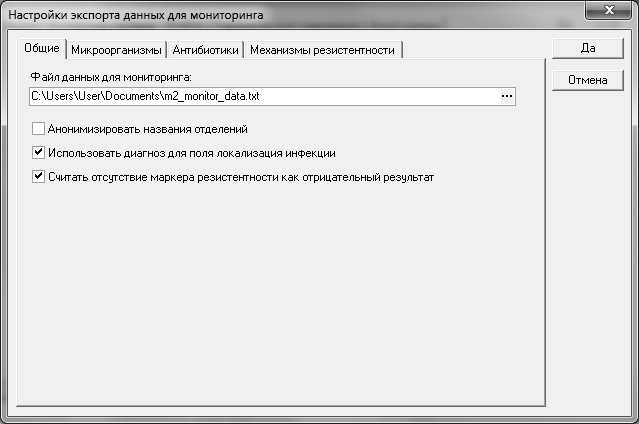
- Файл данных для мониторинга — Задает имя создаваемого файла по-умолчанию. При выполнении экспорта возможно задание другого имени файла.
- Анонимизировать названия отделений — В случае нежелания включать реальные названия отделений, можно установить данный признак. При его установке отделения будут передаваться в обезличенном виде, например: «Отделение#001», «Отделение#002» и т.п.
- Использовать диагноз для поля локализации инфекции — При установке в данном поле будут передаваться названия диагнозов. При снятом признаке – поле локализации будет пустым.
- Считать отсутствие маркера резистентности как отрицательный результат — В программе используются маркеры резистентности значения которых могут передаваться в данные для мониторинга. В отличие от тестов на фенотип, чьи значения также могут передаваться, маркеры резистентности не имеют конкретного значения («отриц.» или «полож.» как тесты на фенотип), поэтому предполагается, что наличие маркера в бланке анализа означает «полож.» результат. При установке данного признака включается следующая логика: если в анализе отсутствует конкретный маркер, но при этом выделен микроорганизм, для которого возможна установка данного маркера, то считаем что данный маркер имеет «отриц.» значение. И это значение будет использовано при формировании файла AMRcloud.
Микроорганизмы
На вкладке Микроорганизмы находится таблица настройки соответствий между микроорганизмами Микроб-2 и AMRcloud:
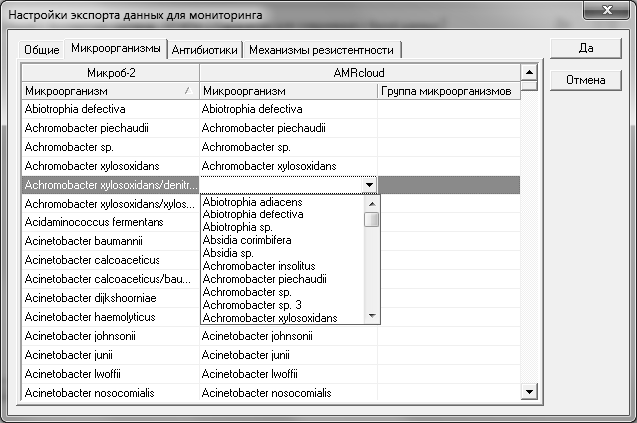
Списки имеют предопределенные фиксированные значения. Установка выполняется путем выбора значения в выпадающих списках для колонок Микроорганизм или Группа микроорганизмов. При невозможности указания конкретного микроорганизма AMRcloud, устанавливайте значение в колонке Группа микроорганизмов.
Антибиотики
На вкладке Антибиотики находится таблица настройки соответствий между антибиотиками Микроб-2 и AMRcloud:
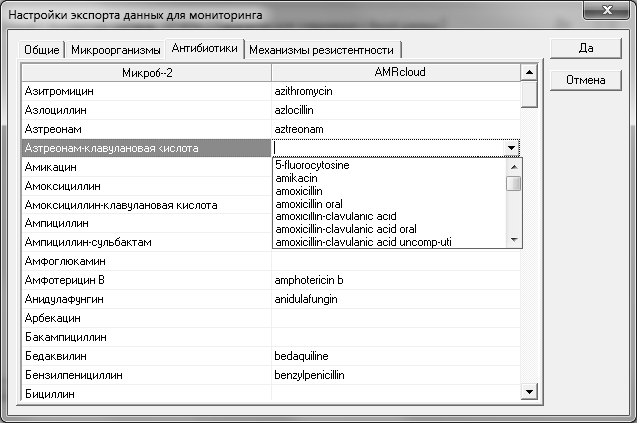
Списки имеют предопределенные фиксированные значения. Для каждого антибиотика Микроб-2 возможна установка только конкретного антибиотика AMRcloud из предопределенного фиксированного списка.
Механизмы резистентности
На вкладке Механизмы резистентности можно настроить передачу той или иной категории механизма резистентности, принятой в AMRcloud:
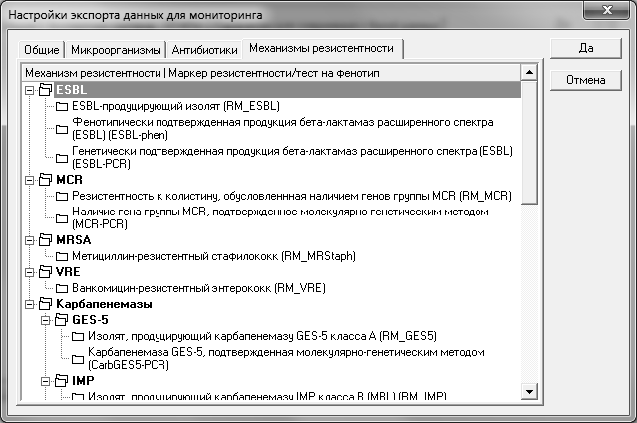
Названия категорий в корне списка. Для формирования результатов механизмов резистентности используются значения тестов на фенотип и наличие/отсутствие маркеров резистентности Микроб-2. Каждой категории механизма резистентности AMRcloud ставится в соответствие набор тестов на фенотип и/или маркеров резистентности Микроб-2. Выбор установки тестов на фенотип или маркеров резистентности через контекстное меню:
Например, редактор списка тестов на фенотип:
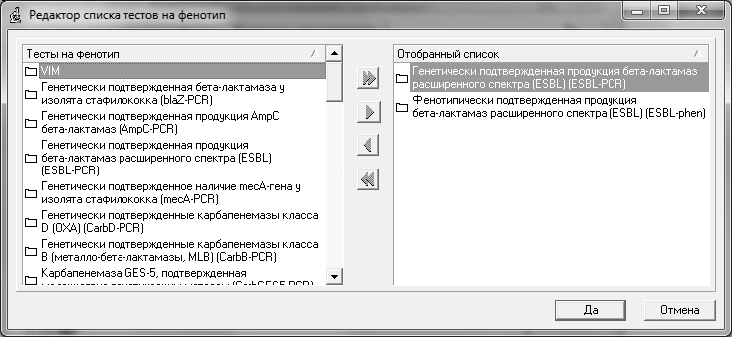
Отобранный список содержит тесты на фенотип или маркеры резистентности для той или иной категории механизма резистентности AMRcloud.