Экспорт из VITEK 2 в AMRcloud
Руководство «Экспорт из Vitek в AMRcloud» рассказывает о том, как имеющиеся у вас данные из анализатора Vitek 2 подготовить для загрузки в онлайн-платформу AMRcloud.
Как загрузить полученный файл подробно описано в Руководстве «AMRcloud. Импорт данных»
Экспорт из VITEK 2
Экспортировать можно только изоляты, перешедшие в неактивную область.
Время перехода изолятов в неактивную область выставляется в общих настройках. По умолчанию – 14 дней.
Незавершенные изоляты (требующие проверки или ввода информации о микроорганизме или пациенте) не переходят в неактивную область. Их необходимо перевести в статус Завершенные.
В главном окне программы нажать на кнопку Инструменты и выбрать Экспорт неактивных изолятов.

В открывшемся окне выставить диапазон дат, поставить галочки на МИК, Фенотип, Подробная информация о карте. При необходимости выбрать другие параметры. Нажать на кнопку Экспорт (верхний правый угол диск со стрелкой).
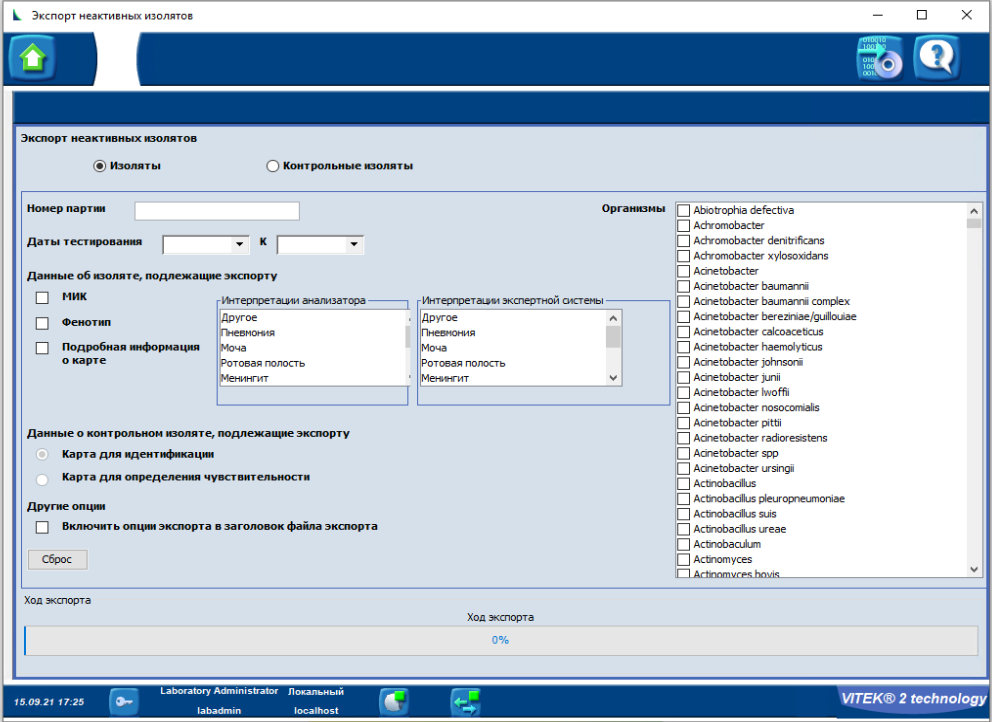
В открывшемся окне нажать Да.
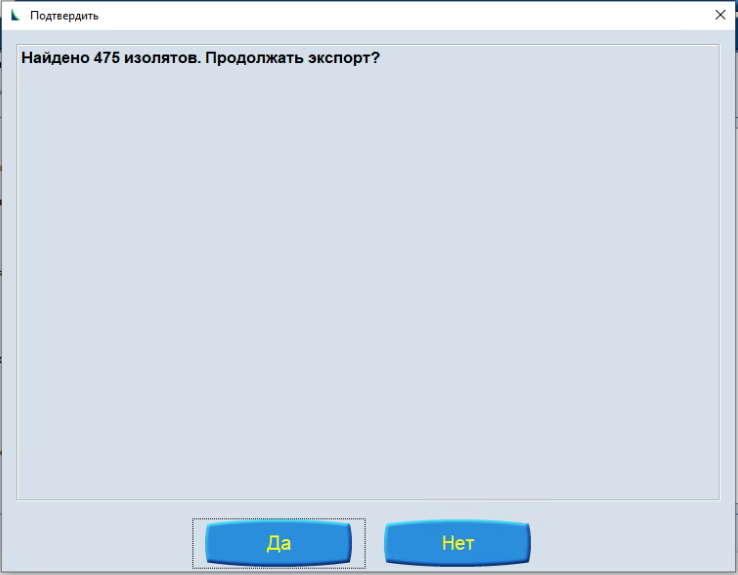
В диалоговом окне выбрать расположение и ввести имя файла. Нажать Экспорт неактивных изолятов.
Дождаться сообщения об окончании экспорта.
Полученный файл необходимо пропустить через конвертер в AMRcloud.
Онлайн-конвертер
Подготовка
Файл с изолятами из VITEK 2 представляет собой текстовый файл (*.txt), разделитель полей - запятая.
Для конвертации файла, перейдите по ссылке https://public.amrcloud.net/vitek.
Загрузка файла
Для загрузки файла нажмите на кнопку Choose file и выберите ваш файл VITEK 2.
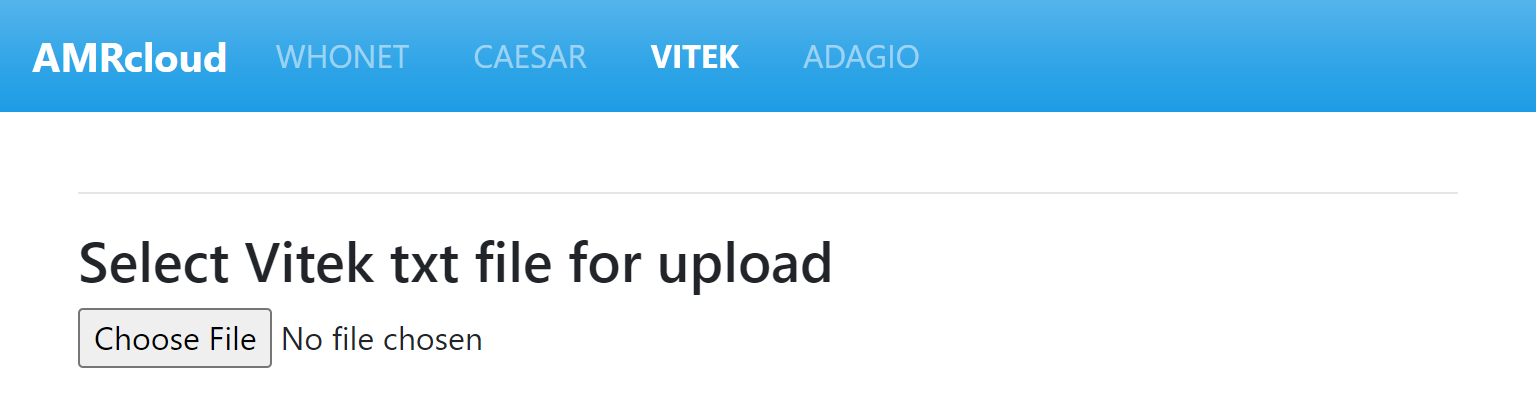
Загрузка файла начнется автоматически. По окончании загрузки система проверит содержимое файла, попытается его прочитать и выведет предпросмотр на экран. В случае успешной загрузки вы увидите следующее.

Если вы видите таблицу и кнопку Convert, значит файл распознался системой.
Конвертация
Процесс конвертации файла заключается в замене кодов антибиотиков и организмов VITEK 2 на обозначения, понятные AMRcloud.
В результате к таблице добавляются следующие столбцы:
AMRCLOUD_ID- искусственно созданный уникальный идентификатор строки в таблицеAMRCLOUD_ORGANISM_ID- перекодированные названия видом микроорганизмов, которые корректно воспримет AMRcloud для простановки интерпретаций данных чувствительности.
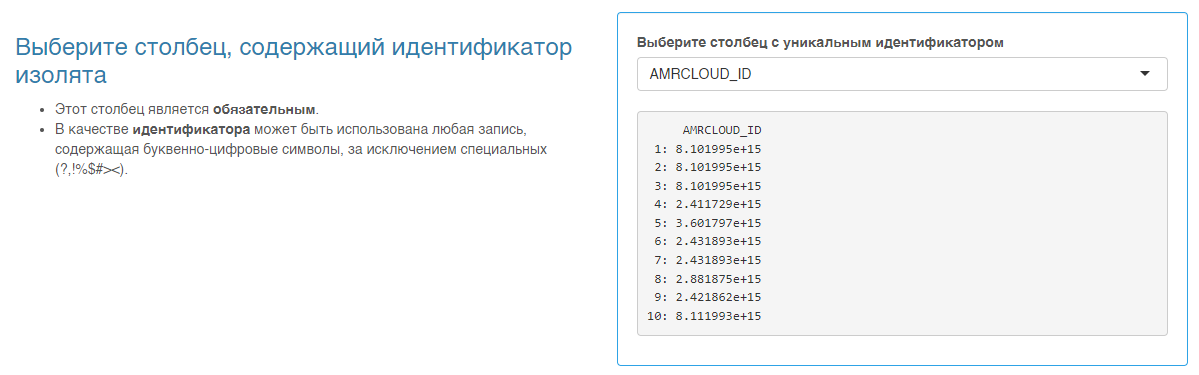
- Для пункта Выберите столбец, содержащий идентификатор изолята
AMRCLOUD_ID - Для пункта Выберите столбец, содержащий названия видов микроорганизмов
AMRCLOUD_ORGANISM_ID

Дополнительно производится объединение значений нескольких столбцов маркеров резистентности в один столбец, начинающийся с кода AMRCLOUD_MARKER_. Также конвертер удаляет столбцы с персональными данными - PATIENT NAME, PATIENT ID, PATIENT LOCATION, LAB ID.
После успешной загрузки файла, нажмите кнопку Convert. По окончании процесса конвертации на странице появится отчет о проделанных манипуляциях. Отчет содержит перечень кодов антибиотиков и организмов, которые были перекодированы, а также сообщения-предупреждения на которые стоит обратить внимание.
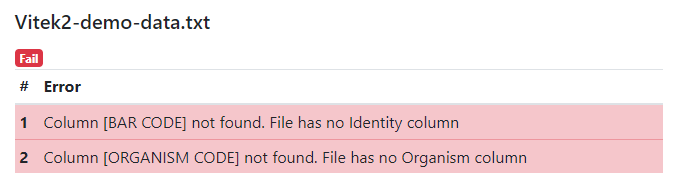
Если при анализе файла возникли ошибки, дальнейшая конвертация невозможна. Необходимо исправить файл.
Возможные ошибки:
- Ошибка Column [BAR CODE] not found. File has no Identity column означает, что не удалось найти столбец BAR CODE с уникальным идентификатором изолята. Необходимо добавить его в исходный файл, либо переименовать существующий столбец с идентификатором.
- Ошибка Column [ORGANISM CODE] not found. File has no Organism column означает, что не удалось найти столбец ORGANISM CODE с кодом микроорганизма изолята. Необходимо добавить его в исходный файл, либо переименовать существующий столбец с кодами.
Существуют следующие виды предупреждений:
- Предупреждение Column [AM-АМПИЦИЛЛИН] looks like antibiotic, but has some non-MIC values выводится в том случае, когда конвертер обнаруживает в столбце с антибиотиком любые нечисловые значения. Название столбца при этом все равно приводится к формату AMRcloud, значения остаются неизменными.
- Предупреждение Column AMRCLOUD_MARKER_АМИНОГЛИКОЗИДЫ added информирует о том, что конвертер обнаружил столбцы со значениями Аминогликозиды и создал новый столбец с объединенными значениями по аминогликозидам.
- Предупреждение Isolate with [BAR CODE]=1234567890 has 2 rows выводится в том случае, когда конвертер обнаружил, что изолят с уникальным идентификатором BAR CODE встречается несколько раз в файле. Рекомендуется исключить дубли из анализа.
- Предупреждение Column [AM-АМПИЦИЛЛИН] has no values выводится когда в столбце с антибиотиком не обнаружено значений.
- Предупреждение Columns with identical names have been enumerated выводится когда при заменах названий столбцов с антибиотиками образовались одинаковые имена. В таком случае столбцы нумеруются по порядку.
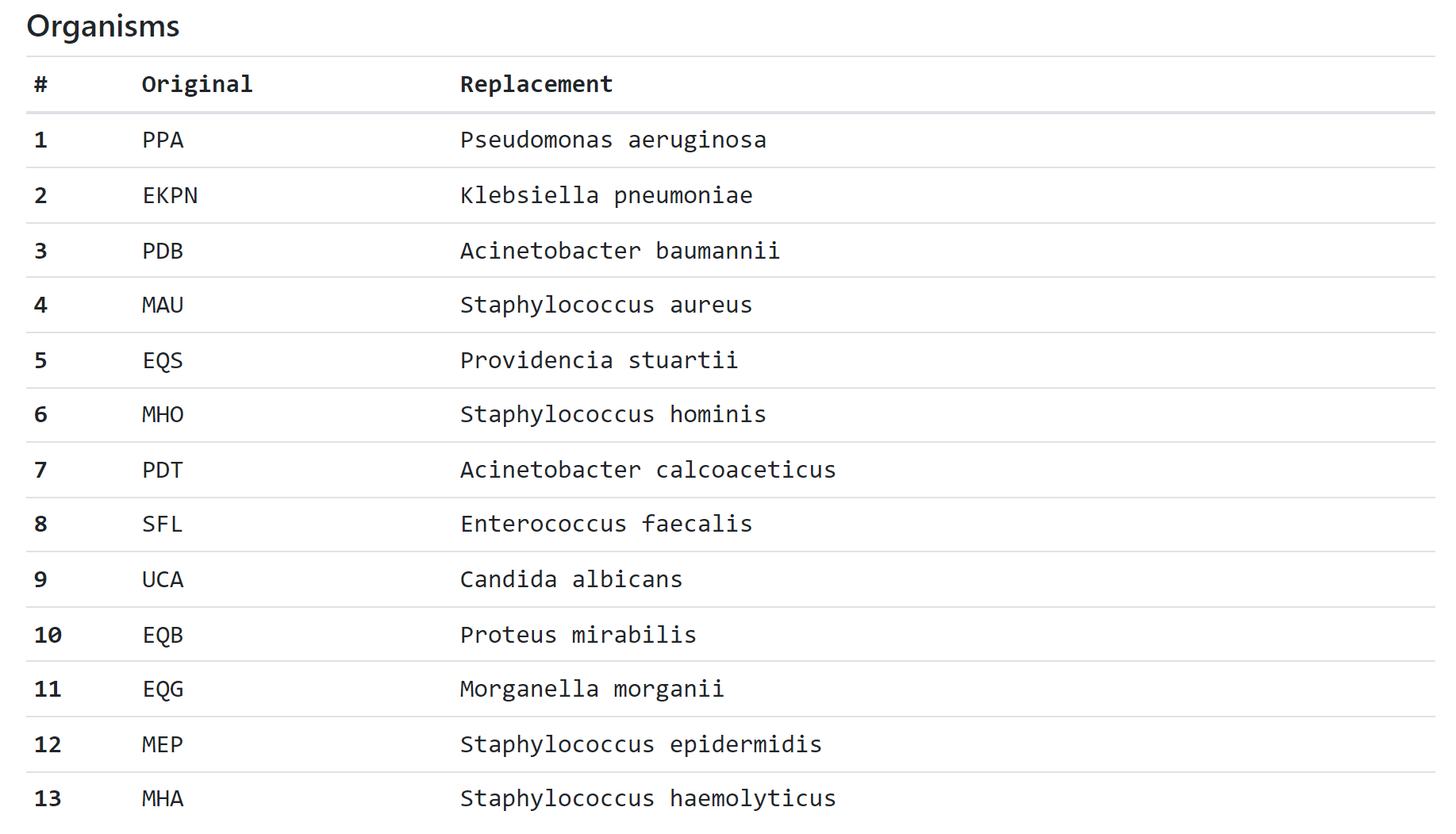
- Предупреждение No translation has been found for organism [CODE] выводится когда конвертеру не удалось сопоставить код микроорганизма из столбца ORGANISM CODE со словарем. В таком случае название данного микроорганизма рекомендуется заменить вручную.


Скачивание файла
Для получения сконвертированного файла, готового к загрузке в AMRcloud, нажмите кнопку Download.
Данный файл может быть использован Вами для создания проектов и наборов данных в AMRcloud. Руководство по загрузке файлов в AMRcloud доступно по ссылке.
Оффлайн-конвертер
Возможна конвертация из формата VITEK 2 в формат AMRcloud и без использования интернета. Для этого необходимо воспользоваться оффлайн-конвертером.
Подготовка
Файл с изолятами из VITEK 2 представляет собой текстовый файл (*.txt), разделитель полей - запятая.
Установка
Для установки программы AMRcloud Vitek Converter скачайте его по ссылке.
Если на компьютере не установлен .NET Framework 4, установщик предложит его скачать. По умолчанию программа устанавливается в папку Документы\AMRsolution\AMRcloud Vitek Converter\
Работа с программой
Окно программы после запуска выглядит следующим образом.
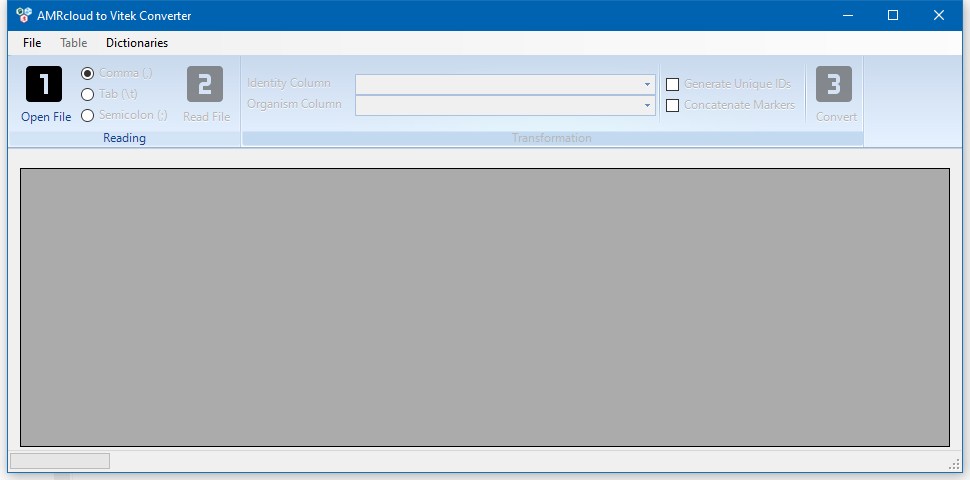
Для выбора файла VITEK нажмите кнопку Open File, либо выберите меню File → Open Vitek File.
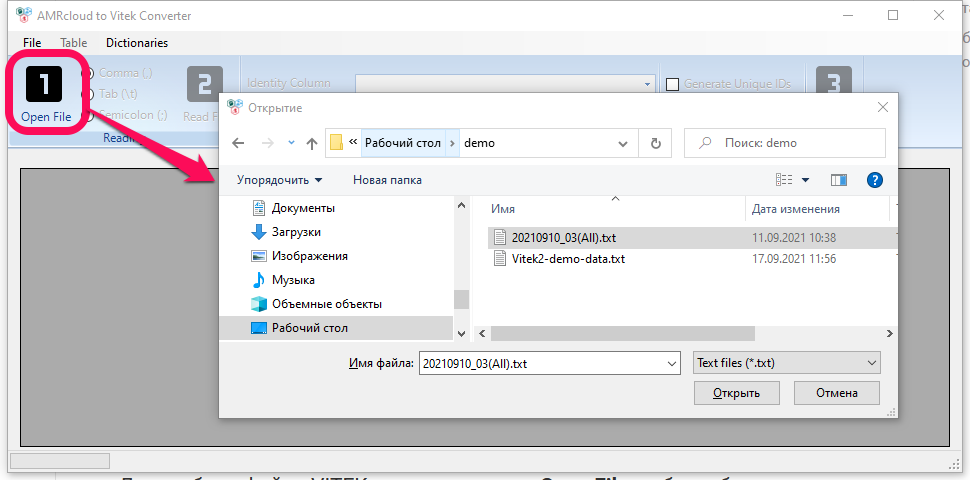
После выбора файла нажмите кнопку Read File, либо меню File → Read Vitek File. Должна появиться таблица. Если таблица не появилась, возможно форматирование файла отличается от стандартного и нужно выбрать другой тип разделителя. Переключатель типа разделителя расположен между кнопками Open File и Read File. По умолчанию используется разделитель запятая.
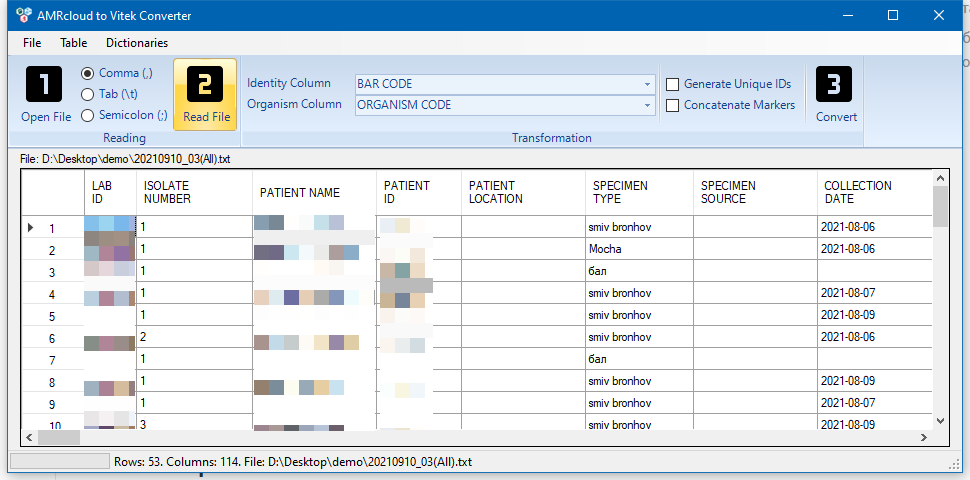
Далее необходимо указать, в каких столбцах содержится идентификатор изолята и код организма. По умолчанию считается, чтобы идентификатор хранится в столбце BAR CODE, а код организма в столбце ORGANISM CODE и эти значения менять не требуется.
При желании вы можете задать дополнительные параметры трансформации:
- Флажок Generate Unique IDs позволяет добавить к файлу новый столбец
AMRCLOUD_IDс новым сгенерированным уникальным идентификатором. - Флажок Concatenate Markers позволяет объединенить значения нескольких столбцов маркеров резистентности в один столбец, начинающийся с кода
AMRCLOUD_MARKER_.
В результате к таблице добавляются следующие столбцы:
AMRCLOUD_ID- искусственно созданный уникальный идентификатор строки в таблицеAMRCLOUD_ORGANISM_ID- перекодированные названия видом микроорганизмов, которые корректно воспримет AMRcloud для простановки интерпретаций данных чувствительности.AMRCLOUD_MARKER_- объединенные значения маркеров резистентности
- Для пункта Выберите столбец, содержащий идентификатор изолята
AMRCLOUD_ID - Для пункта Выберите столбец, содержащий названия видов микроорганизмов
AMRCLOUD_ORGANISM_ID


Для запуска процесса конвертации нажмите кнопку Convert.
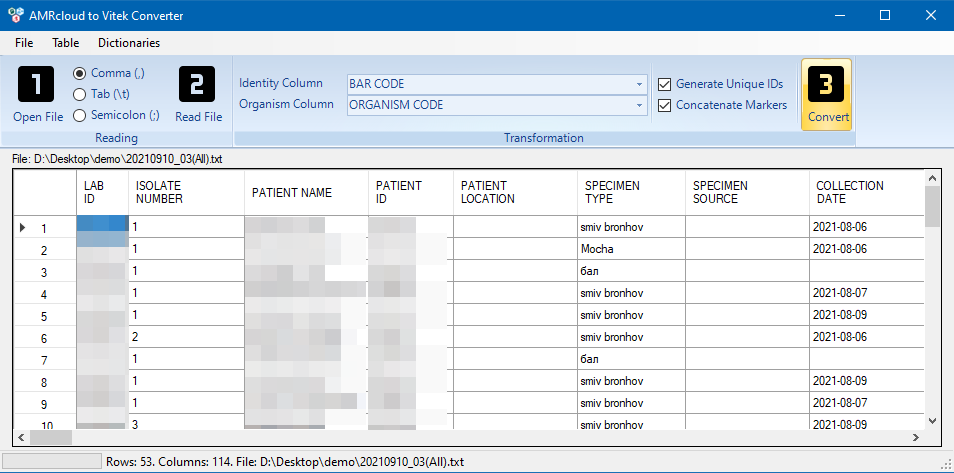
По окончании процесса конвертации на странице появится отчет о проделанных манипуляциях. Отчет содержит перечень кодов антибиотиков и организмов, которые были перекодированы, а также сообщения-предупреждения на которые стоит обратить внимание.
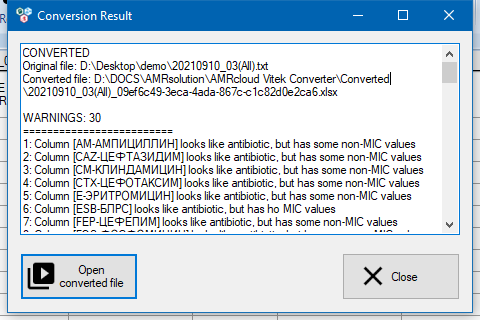
По умолчанию сконвертированный файл сохраняется в папку Документы\AMRsolution\AMRcloud Vitek Converter\Converted. Открыть сконвертированный файл можно с помощью кнопки Open converted file.
Возможные ошибки:
- Ошибка Column [BAR CODE] not found. File has no Identity column означает, что не удалось найти столбец BAR CODE с уникальным идентификатором изолята. Необходимо добавить его в исходный файл, либо переименовать существующий столбец с идентификатором.
- Ошибка Column [ORGANISM CODE] not found. File has no Organism column означает, что не удалось найти столбец ORGANISM CODE с кодом микроорганизма изолята. Необходимо добавить его в исходный файл, либо переименовать существующий столбец с кодами.
Существуют следующие виды предупреждений:
- Предупреждение Column [AM-АМПИЦИЛЛИН] looks like antibiotic, but has some non-MIC values выводится в том случае, когда конвертер обнаруживает в столбце с антибиотиком любые нечисловые значения. Название столбца при этом все равно приводится к формату AMRcloud, значения остаются неизменными.
- Предупреждение Column AMRCLOUD_MARKER_АМИНОГЛИКОЗИДЫ added информирует о том, что конвертер обнаружил столбцы со значениями Аминогликозиды и создал новый столбец с объединенными значениями по аминогликозидам.
- Предупреждение Isolate with [BAR CODE]=1234567890 has 2 rows выводится в том случае, когда конвертер обнаружил, что изолят с уникальным идентификатором BAR CODE встречается несколько раз в файле. Рекомендуется исключить дубли из анализа.
- Предупреждение Column [AM-АМПИЦИЛЛИН] has no values выводится когда в столбце с антибиотиком не обнаружено значений.
- Предупреждение Columns with identical names have been enumerated выводится когда при заменах названий столбцов с антибиотиками образовались одинаковые имена. В таком случае столбцы нумеруются по порядку.
- Предупреждение No translation has been found for organism [CODE] выводится когда конвертеру не удалось сопоставить код микроорганизма из столбца ORGANISM CODE со словарем. В таком случае название данного микроорганизма рекомендуется заменить вручную.
Работа со справочниками
Программа содержит предустановленные справочники антибиотиков и организмов, согласно которым осуществляется конвертация. При необходимости вы можете вносить в них изменения с помощью меню Dictionaries.
Справочники подразделяются на глобальные и пользовательские. Глобальные содержат предустановленные стандартные значения перекодировки. Пользовательские справочники могут переопределять коды глобальных. Поэтому рекомендуется вносить изменения именно в пользовательские справочники.
Работу с ними рассмотрим на примере справочника организмов. Для этого выберите меню Dictionaries → User’s organisms.
Окно справочника выглядит следующим образом.
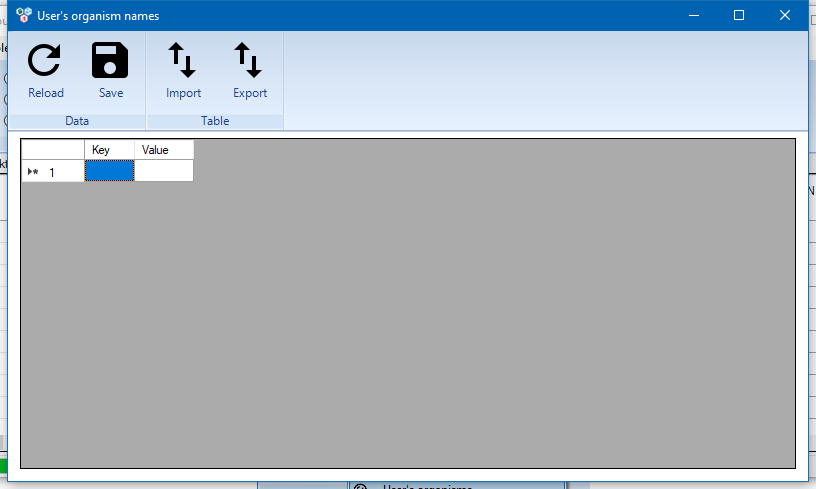
Для ввода данных достаточно дважды кликнуть на ячейку таблицы и ввести текст. Подтверждение ввода осуществляется клавишей Enter. Добавление новых записей осуществляется с помощью строки, помеченной звездочкой. Сохранить изменения можно с помощью кнопки Save. Кнопка Reload позволяет перезагрузить таблицу заново, тем самым сбросить несохраненные изменения.
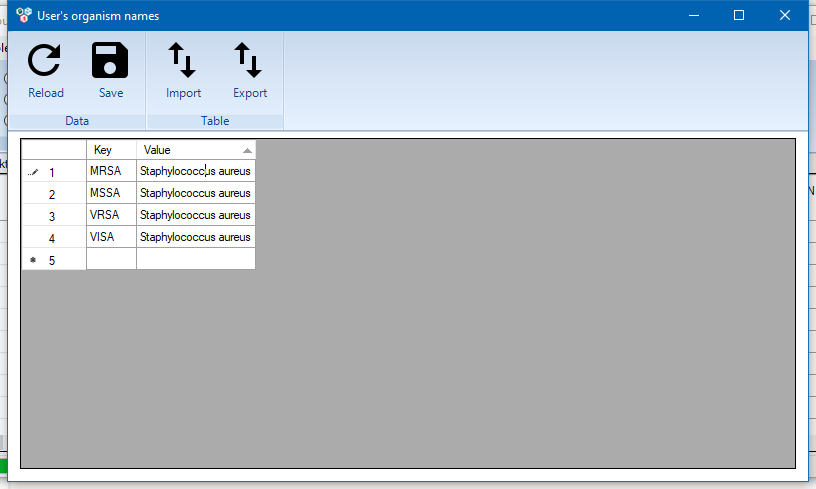
Кнопка Import позволяет упростить заполнение справочника. При нажатии появляется текстовое окно, куда можно скопировать таблицу с кодами из Excel. По нажатии кнопки OK они будут добавлены в справочник.
Кнопка Export позволяет сохранить значения справочника в файл формата Excel.
Справочники Complex antibiotics replacement и Custom antibiotics replacement
Особенность данных справочников в том, что они содержат правила для перекодировки значений МПК для комбинированных антибиотиков.
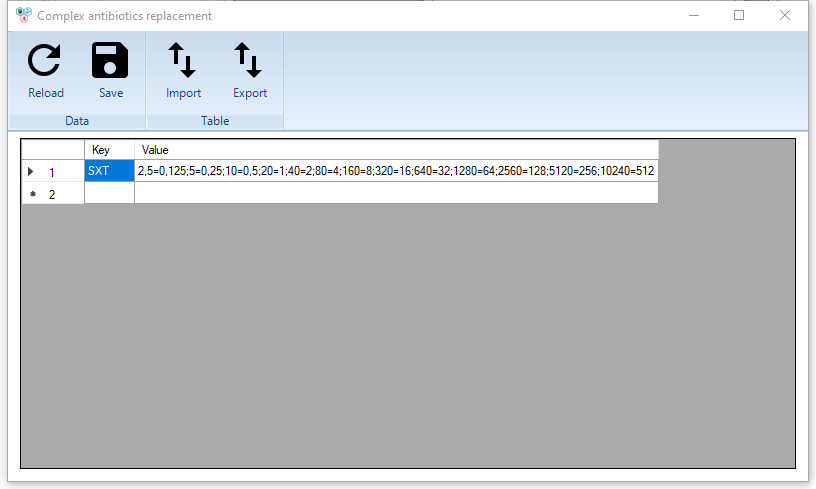
Для описания такого перекодирования необходимо пары значений записать через символ равно (=) , разделителем пар служит запятая.
В качестве примера приводится запись для антибиотика SXT-Триметоприм/cульфаметоксазол. Для словаря использовались следующие справочные данные.
| Vitek | trimethoprim, mg/L | sulfamethoxasole, mg/L |
|---|---|---|
| 2,5 | 0,125 | 2,375 |
| 5 | 0,25 | 4,75 |
| 10 | 0,5 | 9,5 |
| 20 | 1 | 19 |
| 40 | 2 | 38 |
| 80 | 4 | 76 |
| 160 | 8 | 152 |
| 320 | 16 | 304 |
| 640 | 32 | 608 |
| 1280 | 64 | 1216 |
| 2560 | 128 | 2432 |
| 5120 | 256 | 4864 |
| 10240 | 512 | 9728 |