Конвертация из WHONET в AMRcloud
Руководство «Конвертация из WHONET в AMRcloud» рассказывает о том, как имеющиеся у вас данные из программы WHONET подготовить для загрузки в онлайн-платформу AMRcloud.
Как загрузить полученный файл подробно описано в Руководстве «AMRcloud. Импорт данных»
Экспорт из WHONET
Запуск программы
Запустите программу WHONET и выберите вашу лабораторию.
В меню Анализ Данных выберите пункт Анализ Данных.
Тип анализа
Откроется окно подготовки отчета по анализу данных. Подготовка отчета заключается в последовательном задании необходимых условий и опций для создания нужного нам файла.
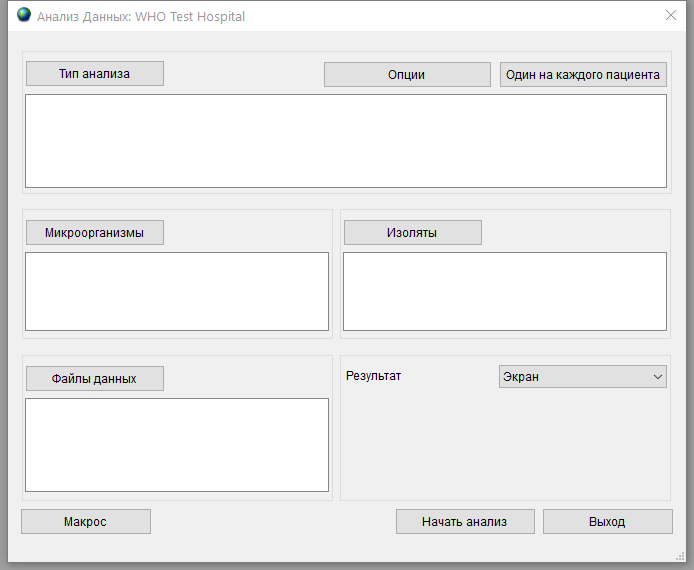
По нажатии кнопки Тип анализа откроется окно выбора анализа. Переходим на вкладку Список изолята и сводка, в разделе Форматы отчетов выбираем 1.Результат загрузки. Нажимаем кнопку ОК.
Дополнительные опции
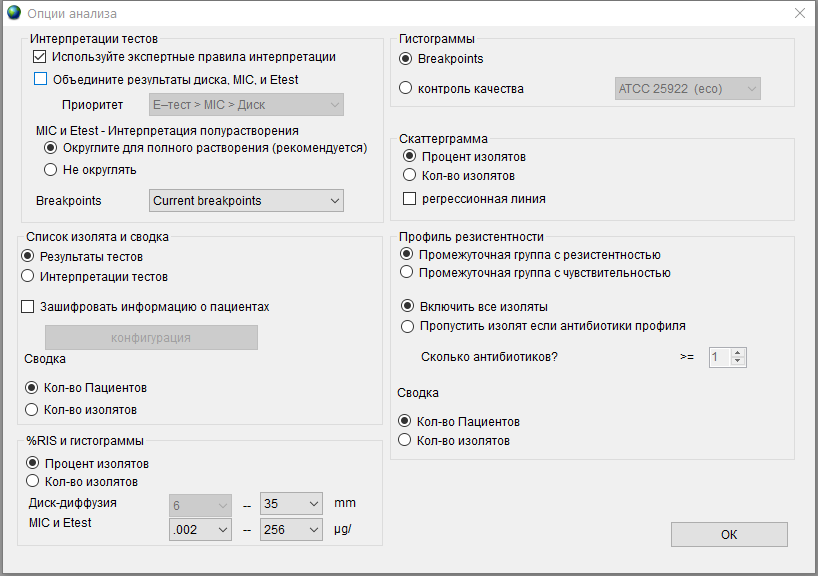
При необходимости можно нажать кнопку Опции и задать некоторые особенности выгрузки результатов. По умолчанию окно Опции анализа выглядит так, как показано на рисунке 5. Таким образом, например, можно объединить результаты тестирования, отметив соответствующую галочку в разделе Интерпретации тестов. Если данные содержат персональную информацию, то полезно будет отметить пункт Зашифровать информацию о пациентах в разделе Список изолята и сводка.
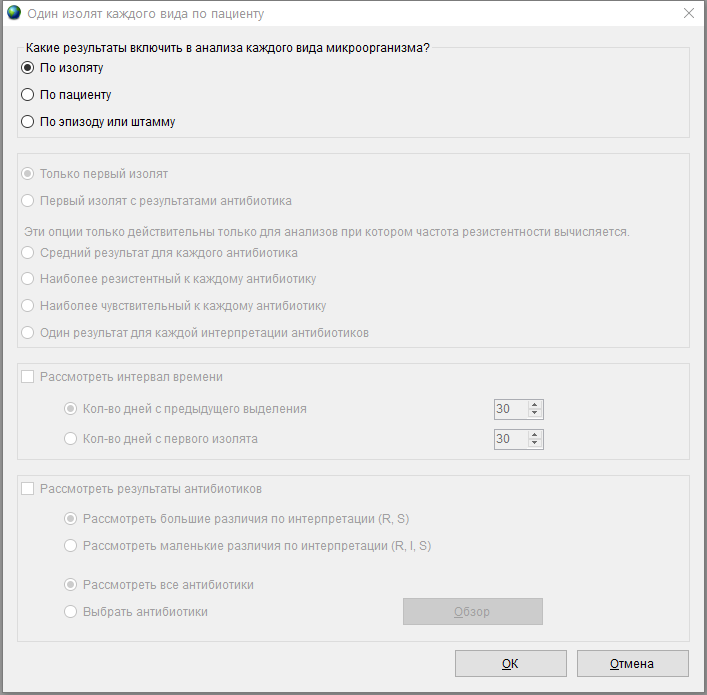
При необходимости, можно изменить условия отбора изолятов в отчет. По умолчанию установлен режим Один изолят каждого вида по пациенту. Чтобы его изменить, нажмите кнопку Один на каждого пациента и отметьте соответствующий режим.
Микроорганизмы
Обязательным пунктом в подготовке файла экспорта является выбор видов микроорганизмов, которые должны попасть в отчет. При нажатии кнопки Микроорганизмы откроется окно Выбор микроорганизма.
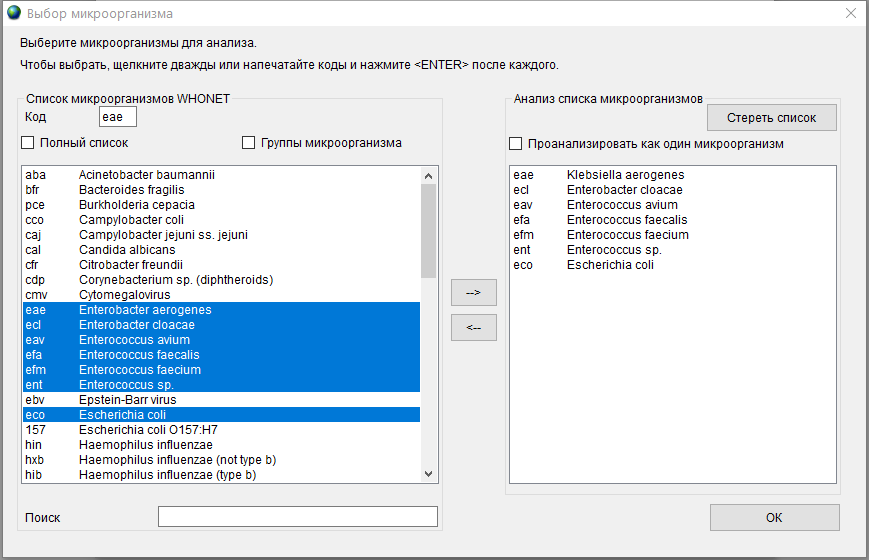
Последовательно кликая по названиям микроорганизмов в левой части окна, соберите необходимый перечень микроорганизмов для включения в отчет. Микроорганизмы можно также выбирать с использованием клавиш Ctrl или Shift. Выделенные микроорганизмы можно добавить в правую часть списка по кнопке –>. Удалить выбранные микроорганизмы можно с помощью кнопки <–.
Изоляты
Дополнительные условия отбора штаммов для включения в отчет можно добавить, нажав кнопку Изоляты. Откроется окно, где можно для каждого из параметров установить критерии выборки.
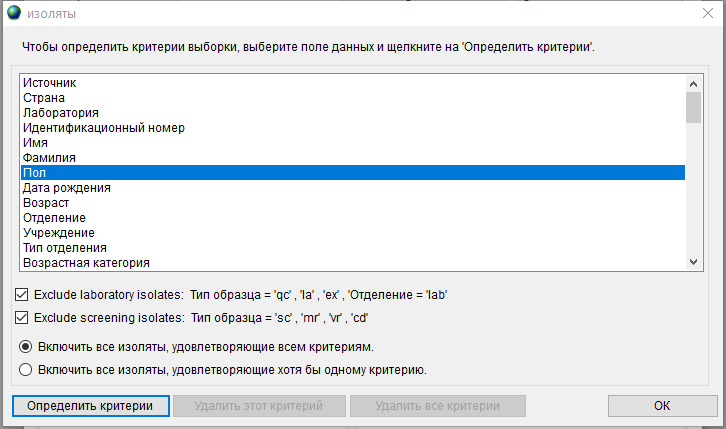
Например, чтобы выбрать изоляты, выделенные только у мужчин старше 30 лет из отделения реанимации, нужно задать три условия:
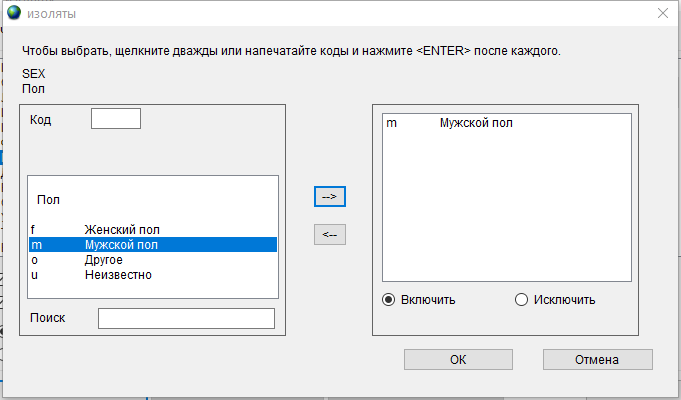
- Сначала создадим критерии отбора по полу. Дважды щелкните по полю Пол, в открывшемся окне выберите m - Мужской пол, нажмите кнопку –> и OK.
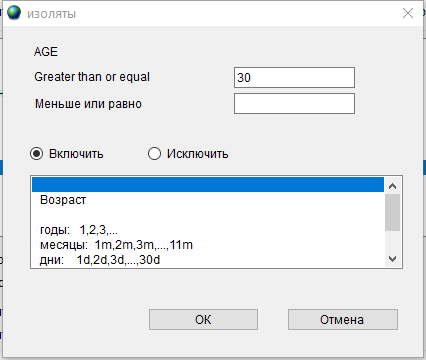
- Затем установим критерий для возраста. Дважды щелкните по полю Возраст, в открывшемся окне в поле Greater than or equal введите значение 30 и нажмите ОК.
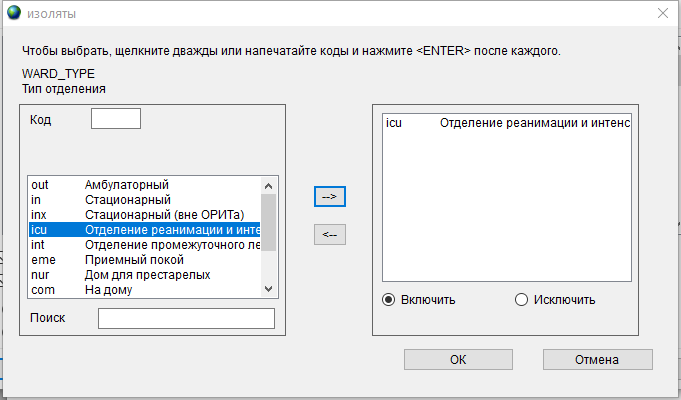
- В качестве финального условия создадим критерий отбора по отделению. Дважды щелкните по полю Тип отделения, в открывшемся окне дважды щелкните по icu - Отделение реанимации и интенсивной терапии и нажмите ОК.
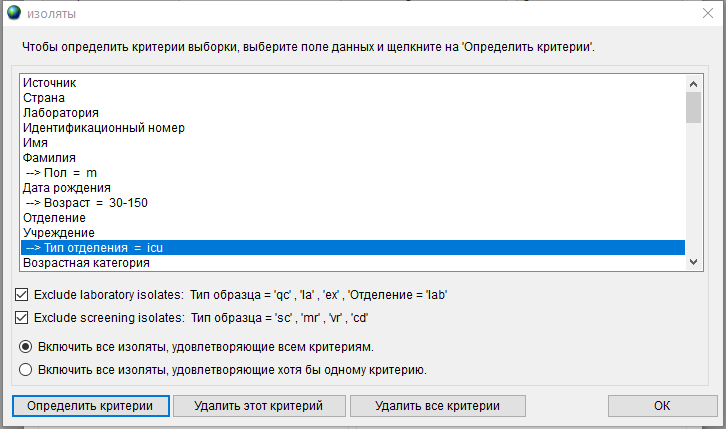
Чтобы все наши три критерия работали вместе, установите переключатель Включить все изоляты, удовлетворяющие всем критериям. Таким образом, окно критериев будет выглядеть следующим образом:
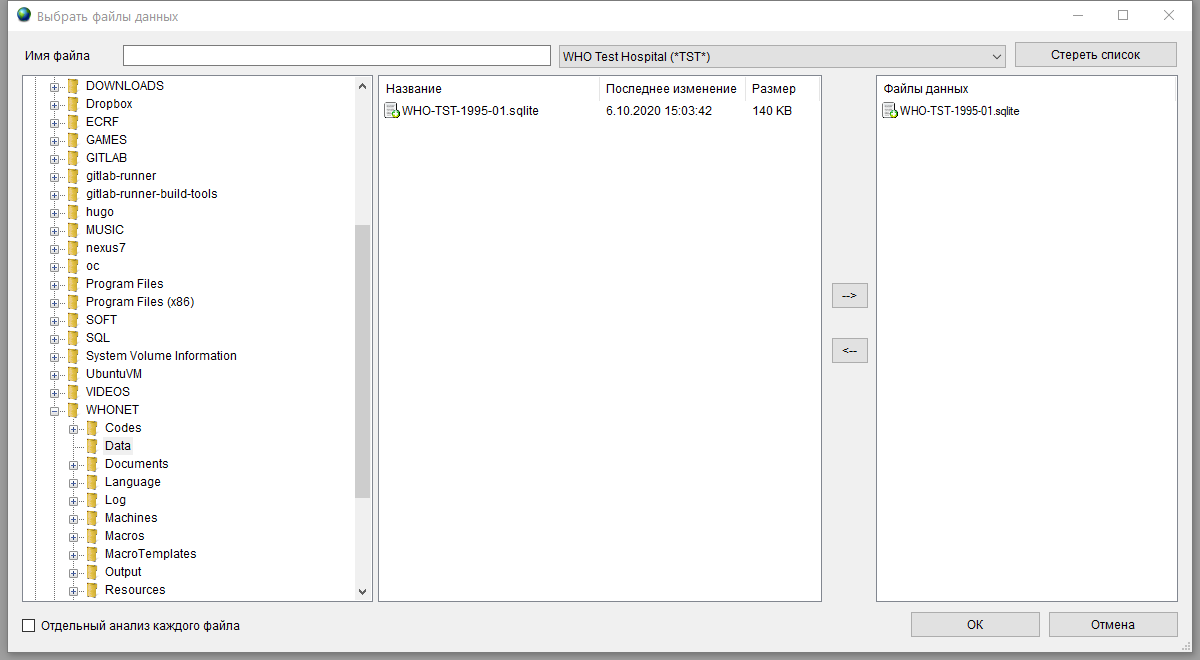
Файл данных
Поскольку все условия отбора и форматирования данных заданы, необходимо указать файл с данными вашей лаборатории. Для этого нажмите кнопку Файлы данных и укажите файл.
Предпросмотр
В результате всех этапов окно Анализ данных примет вид, примерно как на рисунке.
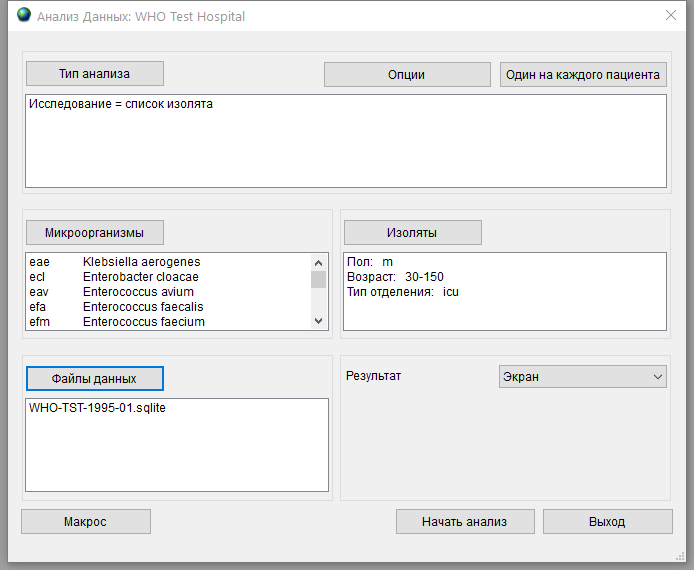
Чтобы предварительно просмотреть, какие изоляты попадут в файл экспорта, выберите в пункте Результат значение Экран и нажмите Начать анализ.
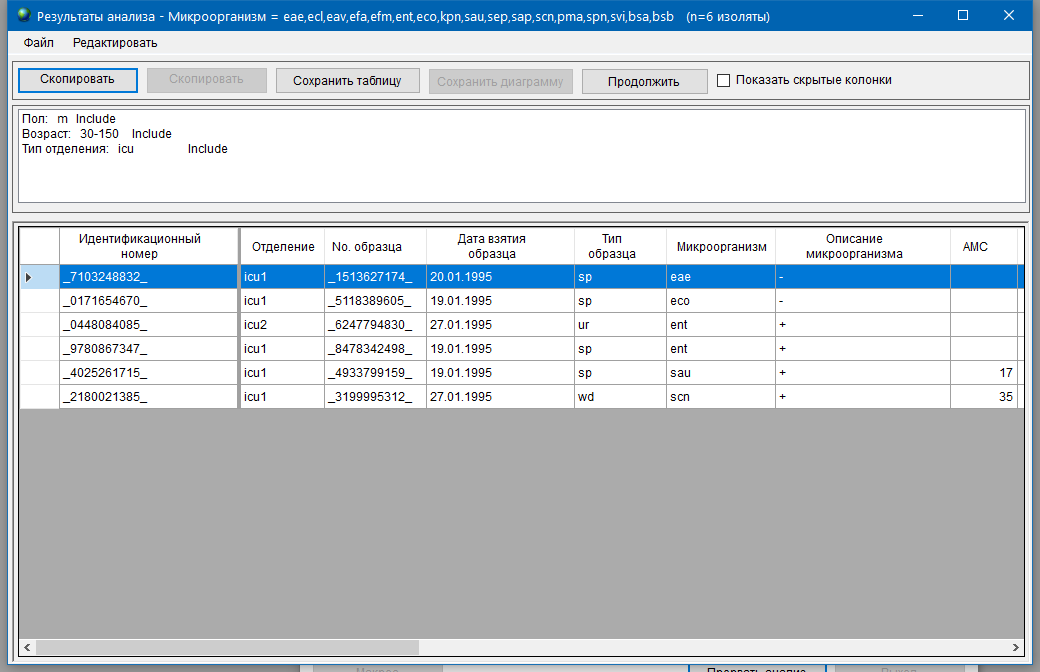
Программа WHONET применит все критерии и условия анализа к вашему файлу данных лаборатории и выведет на экран изоляты, которые удовлетворяют этим условиям. Если результат отличен от ваших ожиданий, то можно изменить условия фильтрации и критерии отбора и повторить вывод на экран.
Экспорт
Для того, чтобы экспортировать изоляты в файл, пригодный для конвертера, необходимо выбрать в пункте Результат значение Текст (WHONET) и задать путь, по которому файл будет сохранен. Окно Анализ данных примет примерно следующий вид.
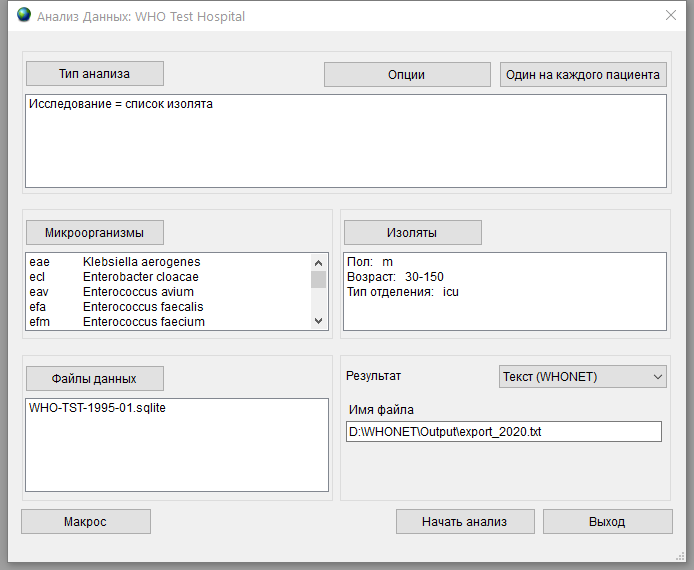
Нажмите кнопку Начать анализ и по указанному пути создастся файл, который будем загружать в конвертер. Если открыть файл в Блокноте, то выглядит он примерно следующим образом.
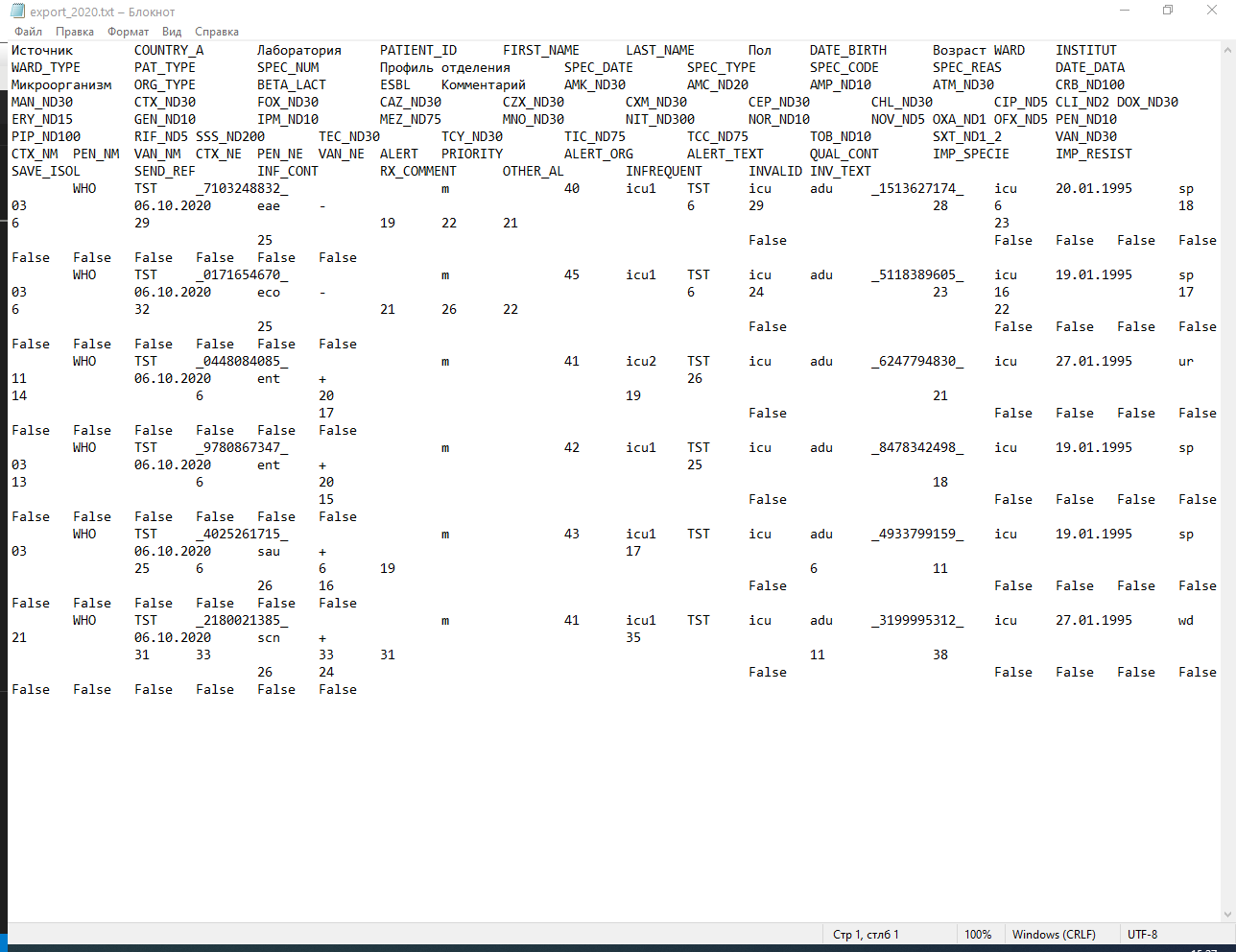
Сохранение настроек экспорта
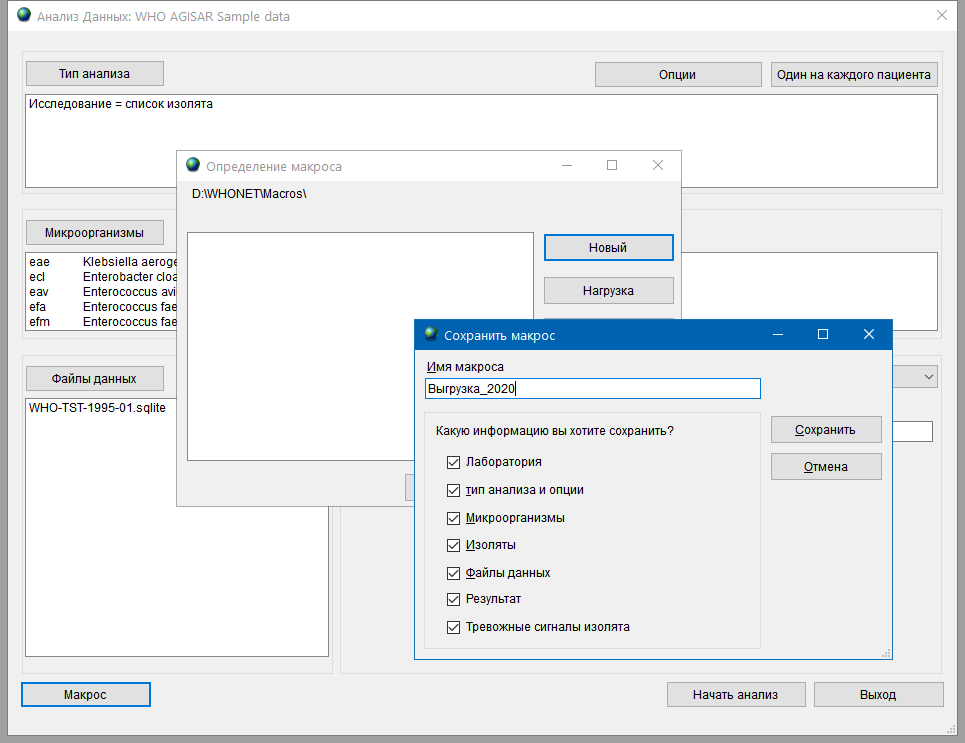
Вполне вероятно, что подобную процедуру придется повторять с некоторой периодичностью. Чтобы каждый раз не выставлять одни и те же значения фильтров, можно сохранить настройки экспорта в специальный макрос. Для этого в окне Анализ данных нажмите кнопку Макрос. В открывшемся окне Определение макроса нажмите кнопку Новый и введите имя файла, в котором будут сохранены настройки экспорта. Дополнительно вы можете отметить галочками, настройки каких разделов требуется сохранить. После этого нажмите кнопку Сохранить и укажите куда сохранить файл макроса.
Чтобы воспользоваться сохраненным макросом, откройте окно Анализ данных, нажмите кнопку Макрос, выберите сохраненный макрос и нажмите кнопку Нагрузка/Load.
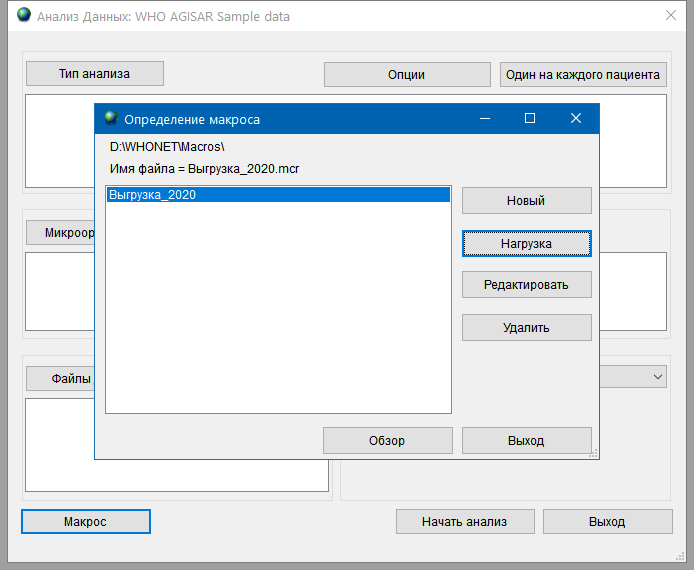
Значения фильтров заполнятся автоматически и после этого достататочно нажать кнопку Начать анализ.
Объединение и экспорт нескольких файлов
В некоторых случаях может возникнуть необходимость экспорта нескольких файлов с данными, например, в разных файлах может храниться информация от разных лабораторий или от одной лаборатории, но за разные года. WHONET позволяет объединить данные из этих файлов и экспортировать их как одно целое.
Для этого в меню Ввод Данных выберите пункт Объединять, экспортировать или шифровать файлы данных.
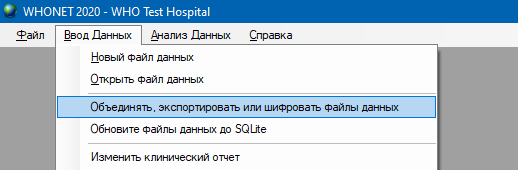
В открывшемся окне выберите файлы, которые необходимо объединить с помощью кнопки Файлы данных. После этого в выпадающем списке Сохранить как выберите формат экспорта WHONET и укажите, куда сохранить новый объединенный файл данных. Для последующей обработки рекомендуется выбрать формат .txt. Если данные содержат персональную информацию, то полезно будет отметить пункт Зашифровать информацию о пациентах.
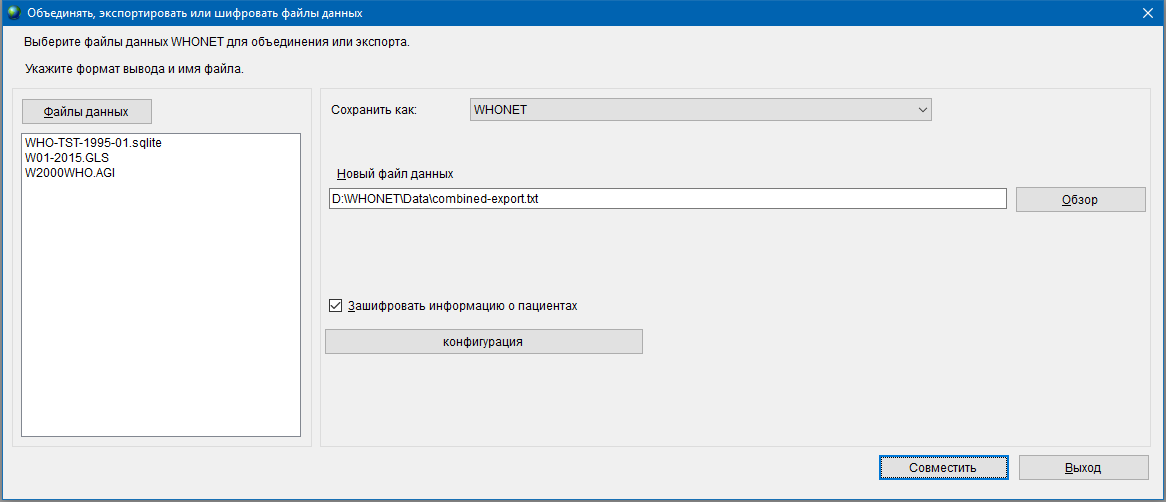
После выполнения всех действий нажмите кнопку Совместить. В результате получится файл, аналогичный полученному с использованием окна Анализ, с той лишь разницей, что разделителем полей является символ вертикальной черты |. Однако все равно данный файл можно загружать в конвертер.
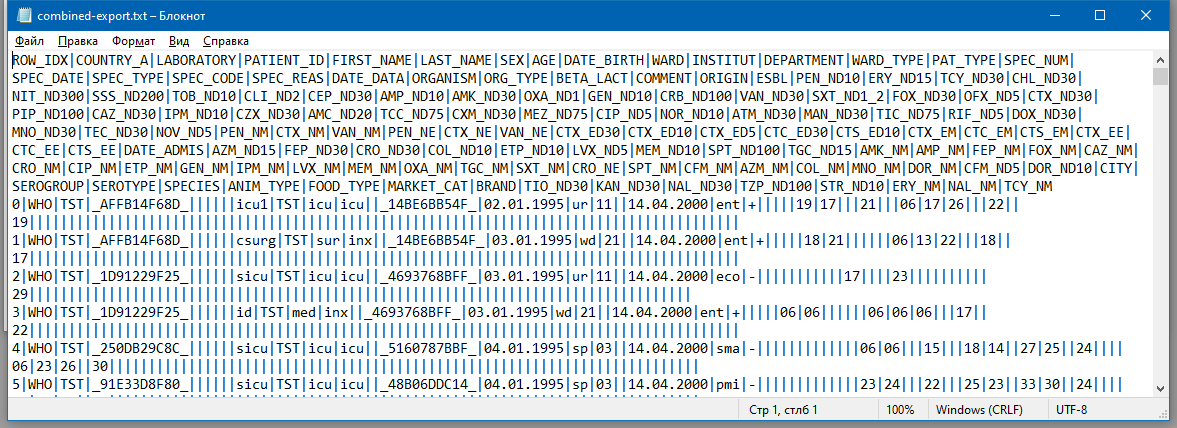
Необходимо отметить, что данное окно можно использовать и для экспорта одного файла — такой способ может оказаться проще и удобнее, если не требуется дополнительных условий отбора изолятов.
Конвертация в AMRcloud
Для конвертации файла перейдите по ссылке https://public.amrcloud.net/whonet.
Загрузка файла
Для загрузки файла нажмите на кнопку Choose file и выберите ваш файл экспорта из WHONET.
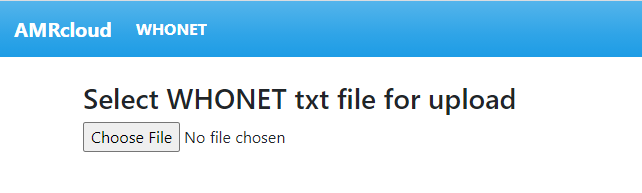
Загрузка файла начнется автоматически. По окончании загрузки система проверит содержимое файла, попытается его прочитать и выведет предпросмотр на экран.
Корректный файл должен удовлетворять следующим требованиям:
- Размер файла не превышает 10 МБ;
- Разделитель полей - табуляция;
- Все имена столбцов уникальные.
В случае успешной загрузки вы увидите следующее.
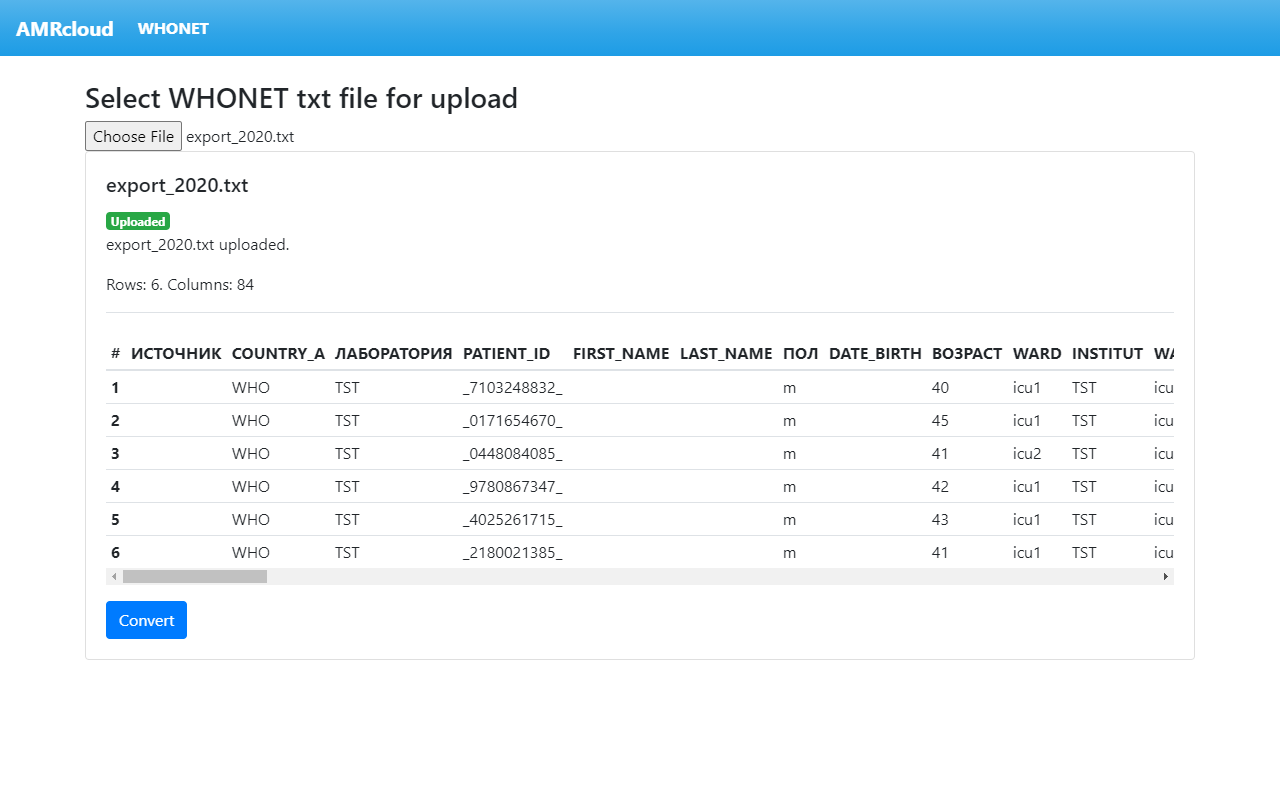
Если же файл прочитался некорректно, вы увидите следующее сообщение:
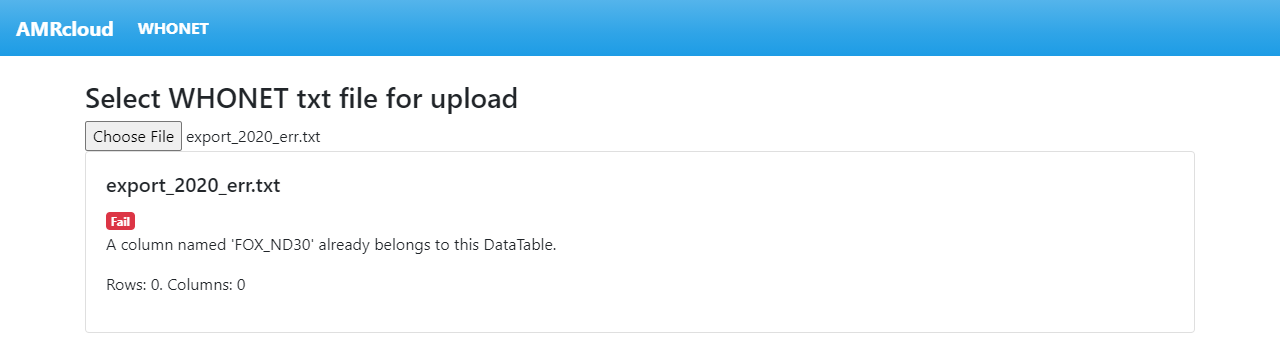
В данном случае ошибка состоит в том, что столбец с именем FOX_ND30 встречается более одного раза в таблице. В случае таких ошибок дальнейшая конвертация невозможна.
Конвертация файла
Процесс конвертации файла заключается в замене кодов WHONET на обозначения, понятные AMRcloud. Такие замены производятся для:
- Антибиотиков (столбцы вида CZX_ND30, VAN_NM, VAN_NE, где первая часть распознается как антибиотик, вторая как метод тестирования)
- Организмов (замена значений столбца ORGANISM/МИКРООРГАНИЗМ, дополнительно добавляется столбец ORGANISM_GROUP)
- Клинических материалов (замена значений столбца SPEC_TYPE)
После успешной загрузки файла, нажмите кнопку Convert. По окончании процесса конвертации на странице появится отчет о проделанных манипуляциях. Рассмотрим пример такого отчета.
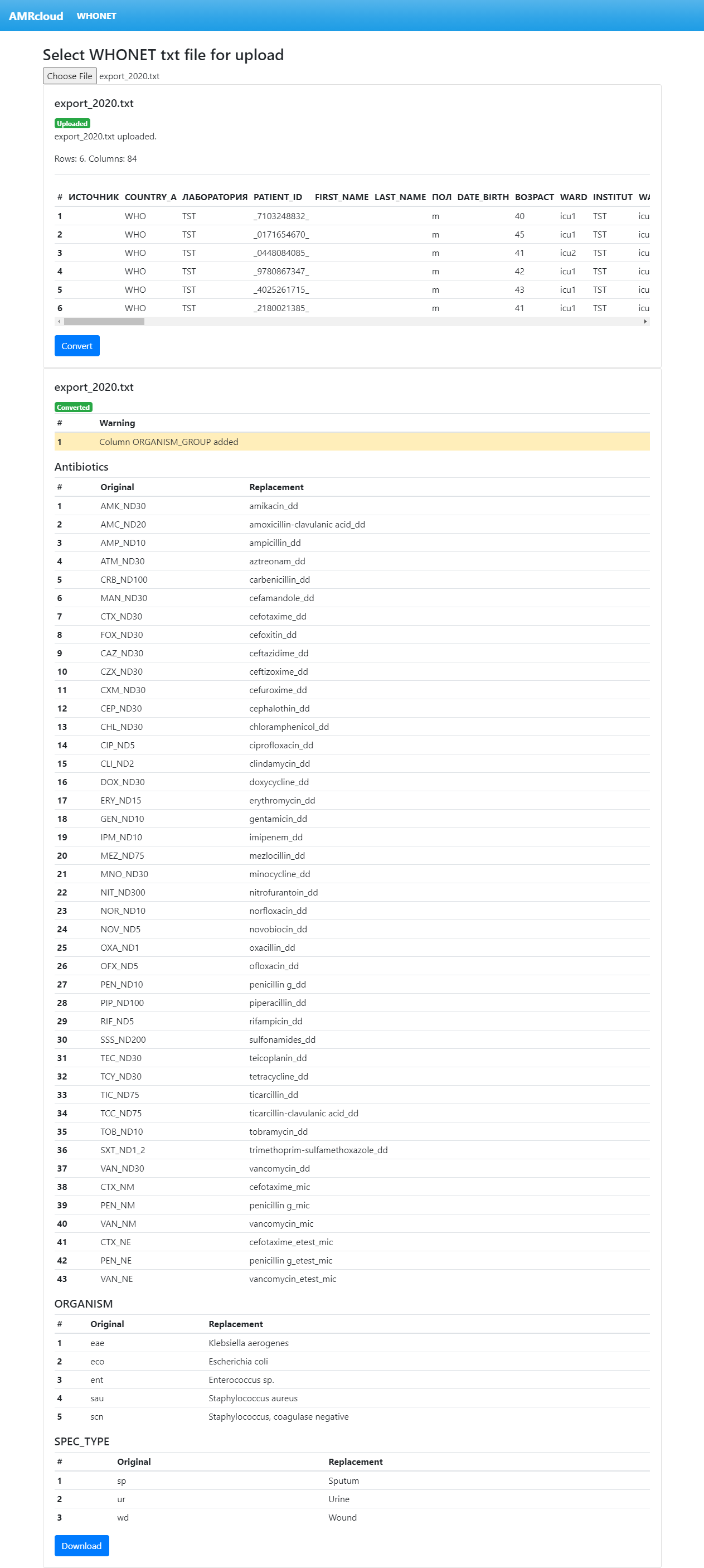
Сначала выводятся предупреждения — это некоторые особенности, выявленные в процессе конвертации, на которые стоит обратить внимание. В данном случае такое предупреждение одно, и оно говорит нам о том, что в исходную таблицу был добавлен и автоматически заполнен новый столбец ORGANISM_GROUP. Это связано с тем, что при загрузке файла в AMRcloud требуется двухуровневая организация микроорганизмов.
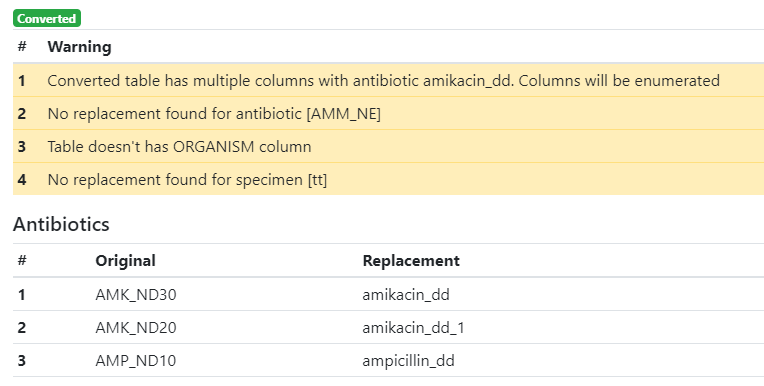
Также возможно появление следующих сообщений в разделе Warnings:
| Сообщение | Комментарий |
|---|---|
| Converted table has multiple columns with antibiotic. Columns will be enumerated | Данное сообщение выводится когда при анализе столбцов с антибиотиками два и более антибиотика в результате перекодировки получают одинаковые замены. Например, в нашем случае в файле было два столбца AMK_ND30 и AMK_ND20. Каждый из них должен заменяться на amikacin_dd, но, чтобы сохранить данные тестирования и уникальность наименования столбцов, к новым именам дописывается цифра. Так AMK_ND30 становится amikacin_dd, а AMK_ND20 – amikacin_dd_1. |
| No replacement found for … | Данное сообщение выводится, когда какое-либо значение, подлежащее замене, не было найдено в словаре. В таком случае сохраняется оригинальное написание. Сообщение выводится для того, чтобы Вы обратили внимание на данное значение и в случае необходимости произвели замену самостоятельно. Например, в нашем случае замены не были найдены для кода антибиотика AMM_NE и материала tt. |
| Table doesn’t has ORGANISM column | Данное сообщение выводится, когда при анализе таблицы, не было найдено столбца ORGANISM/МИКРООРГАНИЗМ. Данная ситуация не является критической ошибкой, а служит сигналом, что конвертированный файл не получится загрузить в AMRcloud без информации о микроорганизмах. |
После предупреждений идут таблицы произведенных замен. Они приводятся для того, чтобы вы могли оценить правильность произведенных замен и, в случае необходимости, исправили некорректные замены самостоятельно.
Отдельно стоит обратить внимание на то, как формируется имя антибиотика на замену:
- Название столбца разбивается на части до и после символа подчеркивания;
- Первая часть заменяется согласно словарю;
- Анализируется содержимое столбца антибиотика. Если находится хотя бы одно значение S, I или R, что дописывается код _sir;
- Если содержимое столбца антибиотика не содержит значений S, I или R, используется вторая часть названия оригинального столбца антибиотика и дописываются коды:
- ND – _dd
- NM – _mic
- NE – _etest_mic
- Если и по анализу второй части оригинального названия столбца антибиотика не удалось определить метод тестирования, то дописывается код _mic.
Скачивание файла
Чтобы скачать сконвертированный файл, нажмите кнопку Download. Предполагается, что файл уже готов для загрузки в AMRcloud и не требует дополнительного редактирования (за исключением ситуации с Warning: Table doesn’t has ORGANISM column, описанной выше).
Как происходит процесс загрузки файла в AMRcloud, можно прочитать в соответствующем руководстве